みなさん、こんにちは。
Folding@Homeは、世界中のPCの演算処理(=分散コンピューティング技術)を用いてタンパク構造を解析するプロジェクトになります。
参考 Folding@Homeのページに飛びますFolding@Homeこの記事では、
- Folding@Homeの仕組み
- Folding@Homeはどういったものを解析しているのか?
- スパコン(=スーパーコンピューター)並みの処理能力が必要になる理由
を調べてみたので紹介したいと思います。
もくじ
Folding@Homeの仕組み
Folding@Homeは分散コンピューティング技術を用い、世界中のPCのパワーを借りて、解析を行っています。
図にすると次のようになります。
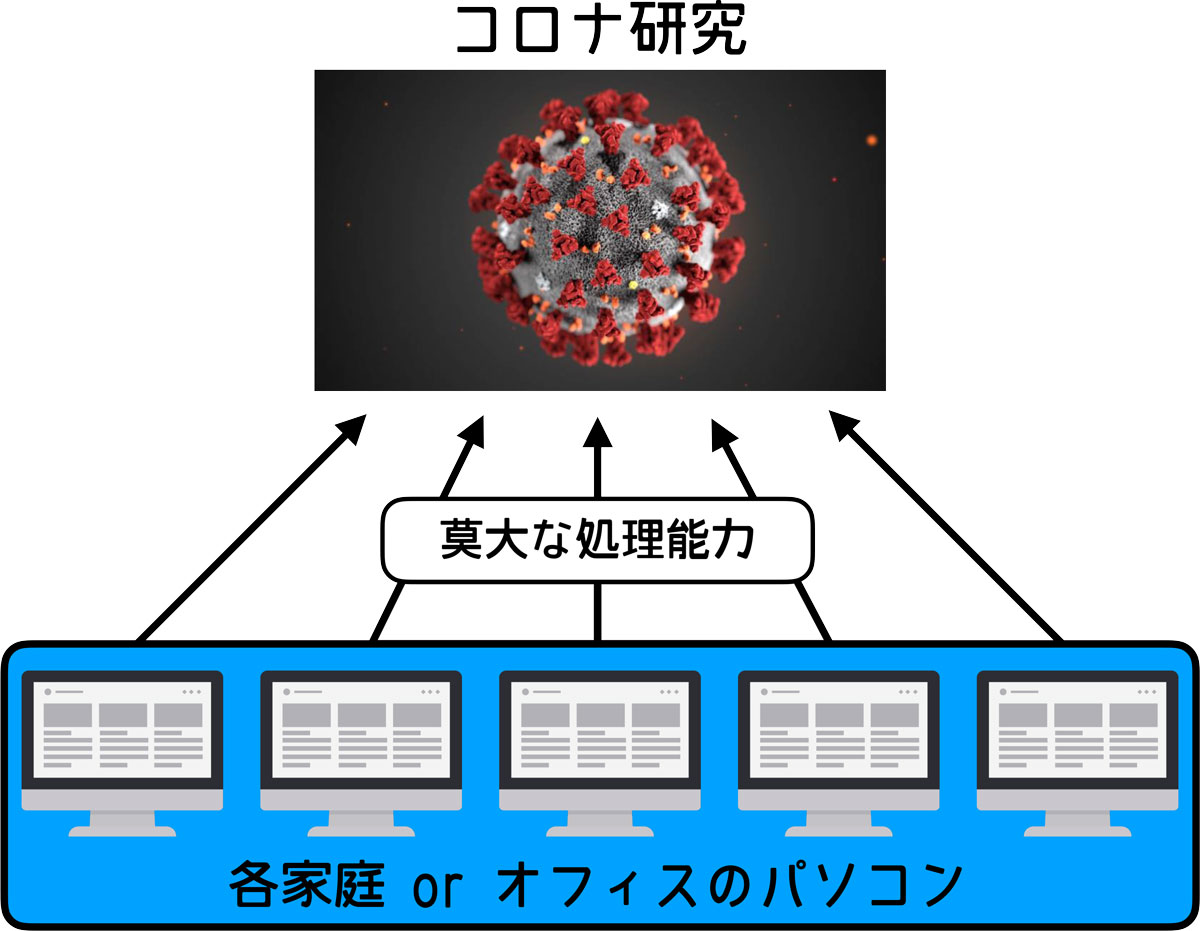
有志の人数が多いほど処理能力は大きくなり、解析処理はどんどん進んでいくというわけです。
どんなメリットがあるの?
- (コロナ)研究が進むことで、研究成果を共有できる
→新型コロナウイルス感染症(COVID-19)に対する治療法が発見できる
新型コロナウイルス(SARS-CoV-2)を解析するプログラムが提供開始となり、参加者が増え、今やFolding@Homeが有する処理能力は世界TOP500のスーパーコンピューターの合わせたものより高いと言われています。
Folding@Homeはどういったものを解析しているのか?

では、そこまでの処理能力を使ってどんなことを解析しているのでしょうか?
PCの処理能力はいくつもの研究に利用されており、その一例を紹介します。
プロジェクトの内容は以下のようになっています。
クリックして内容を確認
The Voelz and Chodera labs have been working with an international team of researchers to computationally screen potential inhibitors of the SARS-CoV-2 coronavirus main protease (also known as SARS-CoV-2 MPro or 3CL). After the virus gets into a cell, it co-opts the cell’s translational machinery and makes a polyprotein (one long chain of amino acids) that needs to be cleaved into smaller pieces that become the functional parts of the virus. The main protease is the viral protein that does the cleavage.
Timely high-throughput crystallization work by the Diamond Light Source XChem project has identified drug fragments that bind the protease, and now the race is on to use these initial hits to drive computational prioritization of compounds to synthesize. We are using the power of Folding@home — now the largest supercomputer in the world — to screen tens of thousands of promising molecules that have been computationally docked to the protease. These include over 6000 compounds designed and submitted as part of the COVID Moonshot challenge.About the calculations
We are using an alchemical free energy calculation method called free energy perturbation, or FEP, to estimate the affinity of a potential drug molecule to its receptor. FEP methods are very expensive compared to other methods like docking, but with the advantages of high accuracy and physical insight. We are using protocols very similar to those used in the most recent SAMPL blind challenge.
The calculation involves decoupling the drug molecule (by turning off its electrostatic and dispersion interactions) from its receptor, and again for a molecule in water. For the latter type of simulations, unfortunately there’s not much to look at the Folding@home viewer, because there’s no protein being simulated. However boring these projects appear, please keep in mind that they are absolutely essential to our success.
この研究では、新型コロナウイルスが宿主の細胞内で翻訳させたポリペプチドをフラグメントに分解するプロテアーゼを阻害する物質をスクリーニングしているそうです。
このスクリーニングには、自由エネルギー摂動法(FEP法) を用いて、新型コロナウイルス由来のプロテアーゼに対する薬物分子の親和性を推定しています。
要するに...
- 新型コロナウイルスが宿主(=人間 etc)の中で作ったタンパク質を機能させるために必要な酵素を阻害する(なおかつ結合のしやすい)薬物分子の探索をしています。
薬物分子が見つかれば、抗ウイルス薬の開発につながります。
その探索(=スクリーニング)に莫大な処理能力が必要となってくるそうです。
スクリーニングにスパコン並みの処理能力が必要になる理由

ではなぜ、スクリーニングにスーパーコンピューター並みの処理能力が必要になってくるのでしょうか?
その答えは、タンパク質の構造にあります。
タンパク質はその複雑さから、折りたたまれて、存在しています。
また、たんぱく折り畳みプロセスは、マイクロ秒(100万分の1秒)単位という高速で、かつ複雑に進むため、一瞬の構造を観察していても意味がなく、ナノ秒単位のシミュレーションが必要になります。
つまり、薬物分子との結合のしやすさ(親和性)を確認するためには、莫大な処理能力が必要となるわけです。
動画に関して
酵素(プロテアーゼ)に結合する薬物分子のシミュレーションはこんな感じで行われているようです。
利用料が高くなってしまうスパコンが、Folding@Homeだとボランティアによるプロジェクトなので、無償(管理サーバー費とかは別にして)になるんですから、素晴らしい試みですよね。
さいごに
未だ新型コロナウイルスに関する解析は終了しておらず、さらなる参加者の募集を呼びかけています。
人類がどれだけ早く、新型コロナウイルスに対する治療法を発見できるかが鍵になるかと思います。
ぜひ、Folding@Homeへの登録して、コロナ研究に協力しましょう!
関連記事
方法-160x160.jpg) 【PC苦手な人向け】Folding@Homeの導入(インストール)方法
【PC苦手な人向け】Folding@Homeの導入(インストール)方法
 【スマホ&PCで研究に貢献】Founding@Home以外にも結構あるんです。分散コンピューティングプロジェクト!
【スマホ&PCで研究に貢献】Founding@Home以外にも結構あるんです。分散コンピューティングプロジェクト!


ってどこ?-160x160.jpg)